
Modulação da microbiota do eixo intestino-pulmão em COVID-19
- 1 Programa de Microbiologia, Instituto de Biociências, Humanidades e Ciências Exatas, Universidade Estadual Paulista (UNESP), São José do Rio Preto, Brasil
- 2 Departamento de Engenharia e Tecnologia de Alimentos, Instituto de Biociências, Humanidades e Ciências Exatas, Universidade Estadual Paulista (UNESP), São José do Rio Preto, Brasil
- 3 Departamento de Bioquímica e Imunologia, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo (USP), Ribeirão Preto (SP) Brasil
- 4 Departamento de Análises Clínicas, Toxicologia e Ciências dos Alimentos, Faculdade de Ciências Farmacêuticas de Ribeirão Preto, Universidade de São Paulo (USP), Ribeirão Preto (SP) Brasil
O COVID-19 é uma doença infecciosa causada pela Síndrome Respiratória Aguda Grave Coronavírus-2 (SARS-CoV-2), e de acordo com a Organização Mundial da Saúde (OMS), até o momento, o SARS-CoV-2 já infectou mais de 91,8 milhões de pessoas em todo o mundo com 1.986.871 mortes. Este vírus afeta principalmente o sistema respiratório, mas o trato gastrointestinal (TGI) também é um alvo, enquanto o SARS-CoV-2 já foi detectado no esôfago, estômago, duodeno, reto e em amostras fecais de pacientes com COVID-19. As manifestações prolongadas do GIT no COVID-19, principalmente a diarreia, foram correlacionadas com a diminuição da riqueza e diversidade da microbiota intestinal, desregulação imunológica e atraso na depuração do SARS-CoV-2. Assim, as interações bidirecionais entre a mucosa respiratória e a microbiota intestinal, conhecidas como eixo intestino-pulmão, devem estar envolvidos nas respostas imunes saudáveis ou patológicas ao SARS-CoV-2. Nesse sentido, a disbiose intestinal está associada ao aumento da mortalidade em outras infecções respiratórias, devido a uma inflamação exacerbada e diminuição dos mecanismos regulatórios ou antiinflamatórios nos pulmões e no intestino, apontando para essa importante relação entre os dois compartimentos mucosos. Portanto, uma vez que as membranas mucosas do trato respiratório e gastrointestinal são afetadas, além da disbiose e da inflamação, é plausível supor que terapias adjuvantes baseadas na modulação da microbiota intestinal e no restabelecimento das condições de eubiose possam ser uma importante terapêutica abordagem para restringir as consequências prejudiciais do COVID-19. Então, nesta revisão, resumimos estudos mostrando a persistência de SARS-CoV-2 no sistema gastrointestinal e as manifestações digestivas COVID-19 relacionadas, além da literatura demonstrando disbiose nasofaríngea, pulmonar e intestinal em pacientes com COVID-19. Por fim, mostramos o potencial papel benéfico da administração de probióticos em outras infecções respiratórias e discutimos o possível papel dos probióticos como terapia adjuvante na infecção por SARS-CoV-2.
Introdução
A microbiota intestinal humana é composta por mais de um trilhão de microrganismos em um ecossistema complexo e dinâmico, regulando o sistema imunológico e toda a nossa fisiologia ( 1 ). Esses micróbios desempenham funções muito importantes no organismo, incluindo metabolismo nutricional, desenvolvimento e modulação da imunidade, bem como defesa contra patógenos nocivos ( 2 ). No trato gastrointestinal (TGI), a barreira epitelial protege contra a invasão de microrganismos patogênicos e ajuda a manter a tolerância aos antígenos alimentares, podendo também estar associada às funções imunológicas sistêmicas e pulmonares. Uma vez danificados, os microrganismos se translocam para a corrente sanguínea ou pulmões e podem induzir septicemia ou síndrome do desconforto respiratório agudo ( 3 , 4)
De fato, há evidências de um crosstalk entre o trato respiratório e o TGI, ou mais precisamente, entre a microbiota intestinal e os pulmões ( Figura 1 ), e essa conexão é denominada eixo intestino-pulmão ( 5 ). Mudanças na composição taxonômica e diminuição da diversidade e função da microbiota intestinal, conhecidas como disbiose, podem afetar a imunidade dos pulmões ( 6 ). Por outro lado, o trato respiratório possui microbiota própria ( 7 ) e a inflamação pulmonar pode levar à disbiose intestinal ( 8 ). Por exemplo, pacientes com infecções respiratórias geralmente apresentam disfunções intestinais ( 9), o que fortalece ainda mais a existência de um eixo intestino-pulmão. Conseqüentemente, as interações do sistema imunológico microbiota intestinal-mucosa e o eixo intestino-pulmão têm sido extensivamente estudados e revisados na literatura científica ( 10 - 13 ).
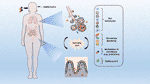
Figura 1 A conexão entre o pulmão e a mucosa intestinal na patogênese da COVID-19. O vírus SARS-CoV-2 infecta preferencialmente células do sistema respiratório, mas um grande corpo de evidências aponta para o GIT como outro alvo importante para a entrada e replicação do vírus. A disbiose, juntamente com o dano da barreira e a inflamação resultante podem facilitar o estabelecimento da doença. A translocação de bactérias, leucócitos e a liberação de mediadores inflamatórios no eixo intestino-pulmão podem contribuir para a deterioração de órgãos associados a COVID-19. Algumas terapias adjuvantes propostas, como prebióticos ou probióticos, que visam restabelecer o estado de eubiose por meio da modulação da microbiota, podem representar uma abordagem alternativa para melhorar ou evitar os piores resultados do COVID-19.
Estudos recentes levantaram a hipótese de que endotoxinas, metabólitos da microbiota, citocinas e hormônios do intestino poderiam atingir a corrente sanguínea e o nicho do pulmão, em um crosstalk bidirecional do eixo intestino-pulmão ( 10 , 13 , 14 ). Além disso, evidências crescentes sugerem uma influência dos metabólitos da microbiota intestinal na migração dos precursores hematopoiéticos da medula óssea e na resolução da inflamação nos pulmões ( 6 , 15 ). Em consonância com isso, o estado imunológico do hospedeiro é influenciado pela microbiota intestinal e pode influenciar a extensão da imunidade a infecções virais, incluindo o SARS-CoV-2 ( 16 , 17) Devido ao seu papel essencial no desenvolvimento e maturação do sistema imunológico, além da indução e regulação das respostas imunológicas nas superfícies mucosas, é plausível assumir que a microbiota e seus metabólitos desempenham um papel significativo na infecção por SARS-CoV-2 ( 13 , 18 , 19 ).
O trato gastrointestinal surge como um alvo para SARS-CoV-2
Como já é amplamente conhecido, está claro que a SARS-CoV-2 afeta principalmente o sistema respiratório. No entanto, a patogenicidade da infecção não se limita exclusivamente aos pulmões; ao contrário, o vírus e a resposta imune subsequente a ele estão relacionados a danos nos tecidos em outros órgãos vitais, enquanto os pacientes em estado crítico apresentam síndromes de disfunção múltipla ( 20 , 21 ).
Vários vírus, como coronavírus, rotavírus e norovírus são capazes de infectar os enterócitos do TGI. A infecção prejudica o processo de absorção causando desequilíbrio na função intestinal ou ativação do sistema nervoso entérico, levando a sintomas e manifestações clínicas importantes da doença ( 22 - 25 ). Em relação à SARS-CoV-2, o GIT também é alvo de infecção e o vírus pode ser detectado no esôfago, estômago, duodeno e reto, além de amostras fecais de pacientes com COVID-19 ( 26 - 28 ). Os principais sintomas gastrointestinais comumente vistos durante COVID-19 são falta de apetite, náuseas, vômitos, diarreia e dor abdominal ( 29 , 30) Nos últimos meses, muitos pesquisadores têm mostrado repetidamente que o SARS-Cov-2 pode, de fato, infectar o GIT ( Figura 1 ) e que há uma alta carga de vírus replicantes, principalmente nas células epiteliais intestinais, conforme observado em biópsias. do intestino delgado e grosso de pacientes infectados ( 31 ). Além disso, a identificação de vírus infecciosos em amostras fecais de pacientes com COVID-19 sugeriu que o GIT poderia ser um local de atividade e replicação viral ( 32 , 33 ). Assim, em um trabalho realizado em Cingapura, 50% dos pacientes positivos para infecção por SARS-CoV-2 tiveram detecção do vírus em suas fezes. Ainda assim, metade deles experimentou manifestações do TGI, como diarreia ( 34) Em outro estudo, a presença de SARS-CoV-2 foi avaliada em esfregaços de garganta e amostras fecais, durante o curso da infecção. As fezes e esfregaços do trato respiratório foram obtidos a cada 1–2 dias até que dois resultados negativos consecutivos fossem alcançados. Os resultados mostraram que, nessa coorte de pacientes, as amostras fecais permaneceram positivas por aproximadamente 5 semanas após as amostras respiratórias apresentarem resultados negativos para o RNA do vírus ( 35 ). Um estudo semelhante reforçou que 80% de uma coorte de crianças infectadas teve swabs retais virais positivos após o teste do trato respiratório ser negativo ( 36 ). Notavelmente, o SARS-CoV-2 vivo também foi detectado em amostras fecais de pacientes que não tinham diarreia, por microscopia eletrônica ( 37) Além disso, em um estudo de pré-impressão, a análise de biópsias intestinais mostrou uma persistência de longo tempo de SARS-CoV-2 no íleo e duodeno de pacientes após a infecção inicial ( 38 ). Assim, a presença ou persistência do vírus no TGI e nas fezes destaca que o SARS-Cov-2 não se limita aos pulmões e aponta para uma potencial transmissão fecal-oral.
Embora a rota específica pela qual o SARS-CoV-2 infecta o TGI ainda não esteja totalmente elucidada, relatórios recentes indicaram algumas possíveis vias envolvidas nele. A etapa crucial da entrada do vírus no organismo hospedeiro é por meio do receptor celular da enzima conversora da angiotensina 2 (ACE2) ( 39 , 40 ) e o sucesso da infecção também requer a protease transmembrana serina 2 (TMPRSS2), em uma etapa de clivagem do vírus Proteína S na membrana da célula hospedeira, permitindo assim uma fusão viral eficiente ( 41 ). Ambos ACE2 e TMPRSS2 têm coexpressão elevada no epitélio esofágico superior e células da glândula, além de enterócitos absortivos do íleo e cólon de indivíduos saudáveis ou pacientes COVID-19 ( 42 , 43) Além disso, as células epiteliais intestinais humanas (hIECs) podem ser infectadas com sucesso por SARS-CoV-2 e, em seguida, ambas as linhas de células epiteliais intestinais e organoides do cólon humano podem ser alvos potenciais para a replicação do vírus, contribuindo potencialmente para o aumento da viremia e disseminação de SARS- Infecção por CoV-2. É importante ressaltar que os autores também descobriram que hIECs infectados com SARS-CoV-2 são capazes de promover uma forte resposta imune mediada principalmente por tipo III, mas não IFNs tipo I. O pré-tratamento de hIECs infectados com SARS-CoV-2, com IFNs exógenos, leva a uma redução significativa das células infectadas, replicação viral e uma diminuição acentuada na geração de partículas virais infecciosas.22 , 44 ). Assim, embora as principais manifestações da infecção pelo SARS-CoV-2 estejam diretamente ligadas ao sistema respiratório, é necessário observar as alterações do TGI que, embora menos comuns, também aparecem durante a doença ( 45 , 46 ).
Manifestações gastrointestinais em COVID-19
A presença de sinais ou sintomas gastrointestinais durante COVID-19 é relativamente comum. Em uma província chinesa chamada Zheijiang, observou-se que entre 651 pacientes com diagnóstico confirmado de COVID-19, de janeiro a fevereiro de 2020, 11,4% apresentavam pelo menos um sintoma gastrointestinal, sendo a diarreia o mais comum (8,14%), com duração de 1 a 9 dias na maioria dos casos, com duração média de 4 dias ( 28 ). Embora o COVID-19 seja menos frequente em crianças ( 47 ), o percentual de manifestações do TGI nesse grupo de pacientes (13,9%) foi muito semelhante ao de adultos, segundo estudo realizado com 244 crianças na cidade chinesa de Wuhan , entre janeiro e março de 2020 ( 48 ).
Pacientes com sintomas gastrointestinais têm uma taxa significativamente maior de doença hepática crônica do que os pacientes com COVID-19, mas sem manifestações do TGI (10,81% vs. 2,95%) ( 28 ), bem como as transaminases aspartato aminotransferase (AST) (16,5% vs. , 5%) e enzimas alanina aminotransferase (ALT) (20,4% vs. 5,9%), que indicam lesão hepática ( 30 ). Esses indivíduos também são mais propensos a ter complicações de síndrome do desconforto respiratório agudo (6,76% vs. 2,08%), progressão para as formas grave e crítica de COVID-19, necessidade mais frequente de uso de ventilação mecânica e internação em unidades de terapia intensiva (UTI) (6,76% vs. 2,08%) ( 28 ), além do tempo de protrombina prolongado (13,1 vs. 12,5 s) ( 30) Em relação aos parâmetros clínicos, pacientes com sintomas gastrointestinais parecem ser mais suscetíveis a febre, fadiga, falta de ar e cefaleia, que podem ser causadas pelo aumento do desequilíbrio eletrolítico ( 28 ). Em comparação com pacientes com COVID-19 e sem sintomas gastrointestinais, aqueles que apresentam essas manifestações também têm maior probabilidade de receber tratamento com antibióticos, interferons e imunoglobulinas ( 30 ). Por outro lado, pacientes com COVID-19 sem comprometimento do GIT apresentam maior incidência de pneumonia unilateral ( 28 ) e têm duas vezes mais chance de recuperação da doença em comparação com aqueles que apresentam sintomas digestivos (30,4% vs. 60%) ( 30 )
À medida que COVID-19 se torna mais grave, os sintomas gastrointestinais tornam-se mais evidentes ( 30 ). No entanto, em relação aos marcadores relacionados à infecção por SARS-CoV-2, não há diferença significativa na quantidade de procalcitonina, proteína C reativa (PCR) ( 28 ) e indicadores de coagulação (exceto para o tempo de protrombina) entre pacientes com e sem COVID -19 manifestação relacionada ao GIT ( 30 ). Também não há diferença no hemograma total ou na função renal.
Os motivos que explicam essas diferenças observadas no curso da doença entre pacientes com e sem sintomas gastrointestinais não estão totalmente esclarecidos, mas é possível que uma replicação viral no trato ( 30 ) possa levar a um quadro clínico mais grave. Além disso, pacientes com sintomas extrapulmonares não clássicos de COVID-19 demoram mais para procurar ajuda médica, facilitando o aumento da gravidade da doença e dificultando a recuperação ( 30 ). Aparentemente, não há diferença na distribuição por gênero dos pacientes com COVID-19 que apresentam sintomas gastrointestinais ( 46 ).
Conforme descrito anteriormente, a ACE2 é a porta de entrada do SARS-CoV-2 na célula hospedeira ( 39 ) e há alta expressão desse receptor no intestino ( 49 ), além da mucosa oral e nas células epiteliais da língua ( 50 ), reforçando assim a ideia de que o GIT também é um alvo importante para a infecção do vírus. De fato, a cavidade oral e o trato digestivo podem servir como uma rota de infecção para a SARS-CoV-2 e a expressão da ECA2 no GIT poderia explicar a presença de sintomas gastrointestinais em pacientes com COVID-19 ( 46 ). Além disso, ACE2 pode controlar a inflamação intestinal e diarreia ( 51); assim, a interação entre SARS-CoV-2 e ACE2 pode levar a uma desregulação desse receptor e aos sintomas intestinais ( 49 ). Além disso, uma vez que ACE2 foi associado com a captura de aminoácidos da dieta, regulação da expressão de peptídeos antimicrobianos e homeostase do microbioma intestinal, é viável assumir que ACE2 pode ser um regulador do microbioma intestinal e da imunidade ( 51 ). De fato, como o SARS-CoV-2 infecta diretamente o TGI, é capaz de gerar uma reação inflamatória que pode levar a danos diretos e indiretos no sistema digestivo ( 30 ).
O uso de antibióticos está associado à diarreia ( 27 ) e o tratamento para COVID-19 pode envolver o uso desses medicamentos ( 29 ), gerando alteração na composição da microbiota intestinal ( 52 , 53 ). Esta poderia ser outra explicação para os episódios de diarreia em pacientes com COVID-19 e reforça a hipótese da relação entre SARS-CoV-2 e a microbiota intestinal ( 54) Assim, é importante manter a vigilância e prestar mais atenção às manifestações do TGI que surgem durante a COVID-19, por serem menos comuns que os sintomas respiratórios clássicos. Além disso, sinais como diarreia não podem ser subestimados devido ao potencial de risco de eliminação do vírus nas fezes e para o diagnóstico precoce de casos suspeitos de COVID-19 ( 28 ).
O Eixo Intestino-Pulmão e a Disbiose em COVID-19
O impacto da microbiota intestinal na imunidade sistêmica e o efeito nas infecções respiratórias foram recentemente explorados em camundongos e humanos ( 14 , 16 , 55 - 59 ). Estudos têm demonstrado o papel essencial da microbiota comensal nas respostas antivirais no pulmão, modulando as respostas imunes na condição homeostática, bem como durante o curso da infecção viral ( 16 , 55 , 60) Notavelmente, os pesquisadores relataram um papel fundamental da microbiota na imunidade antiviral inata no trato respiratório, devido à sua influência nas células epiteliais, macrófagos alveolares e células dendríticas, também modificando as respostas imunes adaptativas celulares e humorais ( 60 , 61 ).
A microbiota intestinal afeta a expressão de receptores de interferon tipo I nas células epiteliais respiratórias, que respondem prontamente às infecções virais por meio da secreção de IFN-α e IFN-β, restringindo a replicação viral ( 60 ). Macrófagos e DCs de camundongos livres de germes não produziram citocinas IFN-α, IFN-β, IL-6, TNF, IL-12 e IL-18 em resposta a ligantes microbianos ou infecção viral e o priming natural killer torna-se defeituoso em a ausência de microbiota intestinal e sinalização de IFN-I ( 56 ). Além disso, usando camundongos tratados com antibióticos, foi demonstrado que a microbiota comensal regula a resposta de IFN-I local e sistêmica por meio da secreção de IFN-β pelas células imunes do cólon. Mais especificamente, o polissacarídeo A capsular derivado de Bacteroides fragilisinduz IFN-β in vitro e em células dendríticas da lâmina própria do cólon em camundongos, sugerindo que a microbiota intestinal pode aumentar a resistência a infecções virais ( 62 ). Além disso, Ichinohe et al. ( 16 ) mostraram que o tratamento com antibióticos e a depleção de bactérias gram positivas do intestino prejudicam a distribuição ou ativação das células dendríticas do trato respiratório e induzem uma diminuição na migração de DCs do pulmão para os linfonodos de drenagem. Além disso, a microbiota intestinal também está envolvida na ativação de células CD4 + e CD8 + específicasLinfócitos T, na expressão estável de pró-IL-1β, pró-IL-18 e NLRP3, enquanto a ativação do inflamassoma favorece a maturação e migração das DCs dos pulmões para os linfonodos de drenagem, após um desafio viral. Abt et al. ( 55 ) relataram uma diminuição da expressão de moléculas de IFN-γRI, MHC-I, CD40 e CD86 em macrófagos peritoneais de camundongos tratados com antibióticos durante uma resposta inicial à infecção viral, sugerindo que os sinais derivados da microbiota intestinal modulam a imunidade inata antes de infecção viral. Em experimentos com um modelo animal MERS-CoV, pesquisadores demonstraram a capacidade do vírus de induzir uma diminuição na expressão de MHC-I e MHC-II em macrófagos e DCs, prejudicando a apresentação antigênica e levando à ativação defeituosa das células T ( 63 ).
Alguns estudos também relataram que os sinais da microbiota comensal exercem diferentes efeitos na mucosa pulmonar, como aumentar o estado antiviral em células epiteliais ou imunes inatas e controlar a replicação viral no início da infecção. A melhora dessa imunidade inata favorece a eficiência das respostas adaptativas celulares e humorais no curso tardio da infecção ( 16 , 55 , 60 ). Assim, podemos supor que micróbios benéficos podem influenciar positivamente o sistema imunológico da mucosa e promover uma resposta eficiente contra os vírus respiratórios ( 64 ) ( 65) A disbiose intestinal está associada ao aumento da mortalidade em infecções respiratórias, provavelmente devido a uma resposta imune desregulada, com aumento da secreção de IFN-γ, IL-6, CCL2 e diminuição de células T regulatórias no pulmão e TGI ( 66 ).
Quatro mecanismos têm sido propostos para explicar o impacto da microbiota intestinal na imunidade da mucosa respiratória: 1) a hipótese de que todos os tecidos mucosos estão interligados, ou seja, a ativação de células imunes em uma mucosa pode influenciar e atingir outros locais mucosos distantes. Assim, a migração de células imunes do GIT para a mucosa do trato respiratório pode estar relacionada aos impactos benéficos exercidos pela microbiota intestinal nas infecções virais respiratórias ( 65 , 67 ); 2) citocinas e fatores de crescimento secretados na mucosa do TGI, em resposta à microbiota comensal, poderiam atingir a circulação sistêmica e atuar em outros tecidos mucosos ( 14 , 65); 3) os padrões moleculares associados a microorganismos (MAMPs) poderiam ser absorvidos e conduzidos para tecidos extra-intestinais, onde ativariam os receptores de reconhecimento de padrões nas células imunes e influenciariam as respostas imunes inatas ( 68 ); 4) os metabólitos da microbiota absorvidos na mucosa intestinal têm sido relacionados à modulação da imunidade mucosa, efeito conhecido como “reprogramação metabólica”. Esses metabólitos, especialmente os ácidos graxos de cadeia curta, ligam-se aos receptores nas células imunes do trato respiratório e aumentam a resposta antiviral no pulmão ( 6 , 64 ).
Com relação ao COVID-19, sabe-se que a infecção das células epiteliais intestinais pelo SARS-CoV-2 pode induzir disbiose, inflamação intestinal e sintomas gastrointestinais ( 31 , 69 ). Além disso, disbiose intestinal prévia observada em diabetes tipo 2, obesidade, hipertensão, doença coronariana e em outros distúrbios relacionados à idade estão envolvidos na desregulação da resposta imune inflamatória ao SARS-CoV-2, favorecendo a infecção, disseminação e gravidade em pacientes com comorbidades ( 70 ). A resposta imune desequilibrada do hospedeiro e a secreção maciça de citocinas inflamatórias, conhecida como "tempestade de citocinas", estão associadas à gravidade da doença e ao pior prognóstico em pacientes com COVID-19 ( 71 , 72) Além disso, um meio inflamatório disbiótico e o dano epitelial induzem a expressão da ECA2, aumentando a replicação do SARS-CoV-2 no GIT e disseminação para outros locais ( 70 ). Isso é consistente com os sintomas gastrointestinais e a detecção de SARS-CoV-2 em esfregaços anais e amostras fecais de pacientes com COVID-19, mesmo nos indivíduos negativos para detecção viral em esfregaços respiratórios ou após a eliminação dos sintomas respiratórios ( 31 , 73 ).
Como já mencionado, vários pacientes com COVID-19 apresentaram sintomas gastrointestinais ( 74 ), achado que poderia afetar potencialmente as interações saudáveis entre a microbiota intestinal e o sistema imune da mucosa, com consequências na resposta imune contra a infecção pulmonar. Além disso, as manifestações prolongadas do TGI, principalmente a diarreia, foram inversamente correlacionadas à diminuição da riqueza e diversidade da microbiota, associada à desregulação imunológica e ao atraso da depuração do SARS-CoV-2 ( 75 , 76) Estudos realizados com pacientes com COVID-19 estão demonstrando que, além da disbiose intestinal, os pacientes podem apresentar desequilíbrio da microbiota faríngea e pulmonar, reforçando a hipótese de que as superfícies mucosas podem estar conectadas e que tudo o que acontece na mucosa do TGI pode ter consequências no outros sites ( 75 , 77 - 81 ).
Em consonância com isso, em estudo realizado na China, Gu et al. ( 75 ) avaliaram a microbiota intestinal de 30 indivíduos COVID-19, 24 pacientes H1N1 e 30 controles saudáveis. Sujeitos infectados com SARS-CoV-2 tiveram diminuição na diversidade da microbiota intestinal quando comparados aos controles, com predomínio de gêneros oportunistas, como Actinomyces, Rothia, Streptococcus e Veillonella , além de diminuição na abundância relativa de micróbios benéficos , como o gênero Bifidobacterium ( Figura 2 ).

Figura 2 Disbiose intestinal e pulmonar em COVID-19. A frequência e a diversidade da microbiota intestinal e pulmonar estão alteradas em pacientes com COVID-19, com predominância dos principais microrganismos bactérias e fungos retratados na imagem. Em contraste, um ambiente homeostático e o estado de eubiose predominam em condições saudáveis.
Da mesma forma, um estudo piloto avaliando a microbiota intestinal de 15 pacientes hospitalizados com COVID-19 relatou alterações significativas durante a internação, com predomínio de microrganismos oportunistas e redução de micróbios benéficos. Mesmo após o desaparecimento do SARS-CoV-2 e a resolução dos sintomas respiratórios, a disbiose intestinal ainda foi detectada. A abundância relativa de Coprobacillus, Clostridium ramosum e Clostridium hathewayi na linha de base correlacionou-se positivamente com a severidade de COVID-19. Além disso, a abundância de Faecalibacterium prausnitzii , que favorece um microambiente antiinflamatório, foi inversamente correlacionada à gravidade do COVID-19 ( Figura 2 ) ( 77) Durante a hospitalização, a abundância relativa de Bacteroides dorei, B. massiliensis, B. ovatus e B. thetaiotaomicron que regulam negativamente a expressão de ACE2 no intestino de camundongo ( 82 ), foi inversamente correlacionada à carga viral nas fezes de pacientes com COVID-19 ( 77 ).
Em um estudo piloto observacional, Zuo et al. ( 79 ) investigaram diferenças no microbioma em 15 pacientes hospitalizados com COVID-19 e sua correlação com o perfil transcricional do SARS-CoV-2. Em 46,7% dos indivíduos, o RNA viral foi detectado nas fezes, mesmo sem manifestações do TGI e após eliminação da infecção respiratória, sugerindo infecção quiescente por SARS-CoV-2 no intestino e real possibilidade de transmissão fecal-oral. Pacientes com elevada infecciosidade de SARS-CoV-2 demonstraram aumento relativo da abundância de Collinsella aerofaciens , C. tanakaei , Morganella morganii e Streptococcus infantis ( Figura 2 ), além do aumento do metabolismo de carboidratos (79 ). Morganella morganii é um micróbio oportunista associado à infecção humana ( 83 ). Pacientes com infecção diminuída de SARS-CoV-2 mostraram prevalência de Alistipes onderdonkii, Bacteroides stercoris , bactéria Lachnospiraceae e Parabacteroides merdae , sugerindo um papel benéfico da microbiota comensal na luta ou competição com o vírus SARS-CoV-2 no intestino ( 79 ).
Em outro estudo do mesmo grupo, Zuo et al. ( 78 ) relataram um aumento na variação interindividual do micobioma fecal em pacientes com COVID-19 em comparação com controles saudáveis. Os pesquisadores avaliaram 30 amostras fecais de pacientes com COVID-19 durante e após a hospitalização e compararam com 30 controles por metagenômica shotgun. Durante todos os momentos de hospitalização, os pacientes com infecção por SARS-CoV-2 apresentaram aumento de fungos oportunistas, incluindo Candida albicans , C. auris , Aspergillus flavus e A. niger ( Figura 2) Esses dois últimos patógenos respiratórios foram detectados em amostras fecais mesmo após a eliminação do SARS-CoV-2 e resolução dos sintomas respiratórios, sugerindo um micobioma intestinal instável e disbiose persistente em alguns pacientes com COVID-19 ( 78 ).
Em relação à microbiota nasofaríngea, De Maio et al . ( 84 ) analisaram amostras de 18 pacientes com COVID-19 leve em comparação com 22 controles não infectados. A microbiota nasofaríngea de pacientes infectados com SARS-CoV-2 e controles foi semelhante, ou seja, não houve diferenças estatisticamente significativas na riqueza e diversidade das amostras coletadas de ambos os grupos, sugerindo uma microbiota resiliente no início de COVID-19 leve. Os principais filos detectados nas amostras foram Firmicutes, Bacteroidetes, Proteobacteria, Actinobacteria e Fusobacteria.
Em um trabalho pré-impresso, Budding et al. ( 80 ) investigaram a microbiota faríngea de 46 pacientes com COVID-19, positivos para detecção de SARS-CoV-2 por PCR, e 89 negativos, e mostraram que existem dois agrupamentos de microbiota diferentes, um agrupamento de microbiota homogêneo com 75% das amostras negativas e outra mais heterogênea com 47% das amostras positivas para SARS-CoV-2. Pacientes mais velhos exibiram diversidade microbiana diminuída e microbiota heterogênea, sugerindo uma dependência da idade na disbiose faríngea e suscetibilidade à infecção por SARS-CoV-2 ( 80 ). Além disso, a microbiota faríngea pode influenciar a progressão das infecções virais respiratórias por meio de vários mecanismos, incluindo a inibição direta da adesão viral e a modulação da resposta imune da mucosa (85 - 88 ). O estado da microbiota faríngea, incluindo a riqueza e diversidade, pode afetar a suscetibilidade à infecção por SARS-CoV-2, a progressão da doença e a probabilidade de infecções secundárias por bactérias patogênicas ( 80 , 89 ).
Os pulmões humanos saudáveis apresentam diminuição da densidade de micróbios, incluindo espécies de Prevotella, Streptococcus , e Veillonella ( 90 - 92 ). Em um estudo observacional, Fan et al. ( 81 ) avaliaram a microbiota pulmonar em biópsias de 20 casos fatais de COVID-19. Acinetobacter, Brevundimonas, Burkholderia, Chryseobacterium, espécies de Sphingobium e membros de Enterobacteriaceae dominaram a microbiota pulmonar nesses pacientes ( Figura 2 ). Família Enterobacteriaceae, que compreende espécies comumente encontradas na microbiota intestinal e inclui alguns micróbios patogênicos, como Enterobacter, Escherichia coli, Klebsiella eProteus , foi detectado nos pulmões de pacientes falecidos com COVID-19 ( 81 ). Dentro do gênero Acinetobacter , o A. baumannii está relacionado a infecções multirresistentes e mortalidade ( 93 ). A microbiota fúngica pulmonar em pacientes com COVID-19 foi dominada por Cryptococcus ( Figura 2 ), seguido por Aspergillus, Alternaria, Dipodascus, Mortierella, Naganishia, Diutina, Candida, Cladosporium, Issatchenkia e Wallemia ( 81 ). As infecções por Cryptococcus foram relacionadas a altas taxas de mortalidade em indivíduos imunocomprometidos ( 94 ), e oIssatchenkia, Cladosporium e Candida representam espécies oportunistas envolvidas em micose em pacientes imunossuprimidos ( 81 ).
Dado o papel crucial da microbiota intestinal na regulação das respostas imunes nas superfícies mucosas e na manutenção da saúde sistêmica e pulmonar, acreditamos que estudos de microbiota são ainda necessários para melhorar nosso conhecimento sobre essas interações no contexto de SARS-CoV- 2 infecção. A identificação das comunidades microbianas da mucosa pode ajudar a encontrar biomarcadores envolvidos na disbiose relacionada à COVID-19, bem como na determinação de potenciais alvos terapêuticos para o desenvolvimento de imunobióticos para o tratamento desses pacientes. De fato, algumas alternativas para a prevenção, diagnóstico, profilaxia e tratamento da COVID-19 já foram propostas, como o uso de inibidores do receptor ACE2 ( 95) e a modulação da microbiota intestinal por meio do uso de probióticos, prebióticos, simbióticos e pós-bióticos, isoladamente ou em combinação, para manutenção do equilíbrio ecológico intestinal, prevenir infecções bacterianas secundárias e também proteger o sistema respiratório ( 47 , 96 ). Essas intervenções terapêuticas também podem melhorar a resposta imunológica em pacientes afetados por comorbidades e, possivelmente, melhorar a imunidade contra o SARS-CoV-2 após futuras vacinações ( 97 ).
Terapias experimentais baseadas na modulação da microbiota
Há um número crescente de estudos avaliando o efeito da administração de probióticos / prebióticos na redução da incidência, duração e gravidade das infecções respiratórias virais em humanos. O potencial para uso de probióticos é apoiado por estudos experimentais, meta-análises e ensaios clínicos sobre o vírus influenza, rinovírus e vírus sincicial respiratório ( 98 - 102 ). Embora os mecanismos não tenham sido determinados na infecção por SARS-CoV-2, algumas cepas probióticas apresentam propriedades antivirais em outros coronavírus ( 103 - 106 ).
Segundo a International Scientific Association for Probiotics and Prebiotics (2013), os probióticos são definidos como “microrganismos vivos que, quando administrados em quantidades adequadas, conferem um benefício à saúde do hospedeiro”. Os probióticos podem ser encontrados em alimentos fermentados e em diversos suplementos, mas apenas cepas bem definidas, com benefício comprovado cientificamente, podem ser utilizadas ( 107 ). O denominado "prebiótico" foi cunhado em 1995 por Gibson e Roberfroid, e a definição atual (2016) é "um substrato que é utilizado seletivamente por microrganismos hospedeiros, conferindo um benefício à saúde", ou seja, a fibra alimentar prebiótica precisa funcionar como substrato para micróbios que promovem a saúde no intestino ( 108) Além disso, simbióticos são definidos como uma “mistura de probióticos e prebióticos que afetam beneficamente o hospedeiro” ( 109 ). Os pós-bióticos incluem substâncias bioativas funcionais resultantes dos processos de fermentação microbiana, incluindo metabolidades como ácidos graxos de cadeia curta e componentes de células bacterianas, que conferem impacto benéfico na saúde do hospedeiro ( 110 , 111 ).
Os probióticos podem ter dois impactos imunomoduladores diferentes no hospedeiro e podem induzir respostas imunes pró ou antiinflamatórias ( 112 , 113 ). Em uma resposta imunoestimulatória, ocorre aumento da atividade fagocítica de macrófagos, células dendríticas e neutrófilos, além de aumento da atividade das células NK, liberação de citocinas inflamatórias e polarização Th1 / Th17 na mucosa intestinal ( 114 - 117 ). Em uma resposta anti-inflamatória, algumas cepas probióticas podem induzir células T regulatórias, via modulação de células dendríticas na mucosa intestinal, induzindo IL-10, TGF-β e aumentando a secreção de IgA e a função de barreira intestinal ( 118 - 120) Portanto, o conhecimento da cepa probiótica e estudos experimentais são essenciais para determinar a melhor cepa para atingir os objetivos terapêuticos. Portanto, uma vez que os probióticos podem modificar o equilíbrio dinâmico entre os mecanismos inflamatórios e regulatórios e impactar a eliminação viral, a resposta imune e o dano pulmonar, seu uso pode ser crucial para atenuar a síndrome do desconforto respiratório agudo e prevenir complicações maiores da SARS-CoV-2 infecção ( 102 , 121 ).
Em modelos experimentais murinos, algumas cepas probióticas de Lactobacillus estimulam a secreção de IFN-γ, IL-6, IL-4 e IL-10 nos pulmões, e uma diminuição em S. pneumonia e sua disseminação para a corrente sanguínea ( 122 ). Além disso, Lactobacillus casei aumenta os processos fagocíticos e de morte em macrófagos alveolares, expressão de IFN-γ e TNF-α, auxiliando no combate ao vírus influenza ( 123 ). Em humanos, um ensaio clínico randomizado usando Lactobacillus plantarum DR7 relatou supressão das concentrações plasmáticas de citocinas inflamatórias, como IFN-γ e TNF-α, e aumento de IL-4 e IL-10 em adultos jovens com infecções respiratórias superiores ( 124) Dada a tempestade de citocinas observada em COVID-19, esta abordagem terapêutica poderia beneficiar os pacientes por mecanismos como o restabelecimento da barreira intestinal por meio do aumento da expressão de tight junctions e aumento da produção de ácidos graxos de cadeia curta, incluindo butirato, que têm efeito antiinflamatório e poderia, teoricamente, reduzir a invasão de colonócitos por SARS-CoV-2 ( 102 ).
Existem vários estudos que mostram o impacto da suplementação de probióticos na prevenção de infecções do trato respiratório superior e inferior em humanos. Em uma meta-análise incluindo 12 ensaios clínicos randomizados e 3.720 indivíduos, a administração de probióticos reduziu o número e a duração dos episódios respiratórios superiores agudos, a duração do antibiótico e a gravidade da doença ( 125 ). Os probióticos também têm sido usados para prevenir infecções bacterianas do trato respiratório inferior em pacientes criticamente enfermos. Duas meta-análises incluindo quase 2.000 pacientes mostraram que a suplementação de probióticos diminuiu a incidência de pneumonia associada à ventilação ( 126 , 127 ).
A senescência imunológica e a diversidade diminuída da microbiota intestinal aumentaram potencialmente a incidência de infecções em idosos, que estão sob risco crescente de COVID-19 ( 128 , 129 ). Assim, a ingestão diária de alimentos fermentados, contendo probióticos, poderia melhorar o desempenho do sistema imunológico por meio da interação com a microbiota da mucosa do TGI. Em um ensaio clínico duplo-cego e controlado, Guillemard et al. ( 130 ) avaliaram o efeito de produtos lácteos contendo Lactobacillus casei em 1.072 indivíduos, com idade mediana de 76 anos, durante 3 meses, e mostraram que os probióticos diminuíram significativamente a incidência e os episódios de infecções respiratórias superiores.
Além de prevenir infecções respiratórias superiores e inferiores, os probióticos podem auxiliar no tratamento da diarreia associada à própria infecção por SARS-CoV-2 ou causada por antibióticos usados para tratar infecções pulmonares secundárias ( 131 , 132 ). Um dos fatores de risco associados à infecção por SARS-CoV-2 é a pneumonia bacteriana secundária. Em trabalhos recentes sobre COVID-19, as infecções secundárias foram significativamente correlacionadas ao pior prognóstico, desfechos e morte ( 81 ). Uma meta-análise realizada por Szajewska et al. ( 133 ) usando 18 ensaios clínicos randomizados controlados, com 4.208 participantes, demonstraram que Lactobacillus rhamnosus por via oralA administração de probióticos GG foi associada à diminuição da duração da diarreia e redução das hospitalizações em pacientes hospitalizados. Os antibióticos induzem alterações significativas no equilíbrio da microbiota intestinal, que podem resultar em diarreia associada aos antibióticos. Os probióticos podem prevenir essa condição por meio do reforço da barreira epitelial e restauração da eubiose. De fato, uma meta-análise incluindo 33 ensaios clínicos randomizados e controlados, com 6.352 indivíduos, demonstrou que a suplementação de probióticos confere um impacto protetor moderado na diarreia associada a antibióticos, reduzindo sua duração ( 134 ).
A melhora da microecologia intestinal e do processo de eubiose pela ingestão de probióticos pode promover um sistema imunológico regulado e prevenir uma inflamação excessiva ou infecções secundárias ( 97 , 135 - 137 ). De acordo, algumas cepas de Bifidobacterium, Lactobacillus paracasei e L. rhamnosus reduzem a ocorrência de infecções respiratórias, como H1N1, H3N2 e H5N1, aumentando as respostas imunes à vacina ( 67 , 138 , 139) Essa melhora nas interações do sistema imunológico microbiota-mucosa também pode beneficiar as respostas imunológicas à vacinação contra o vírus SARS-CoV-2. No entanto, embora levantemos a hipótese de que a disbiose ou a modulação da microbiota podem afetar potencialmente a eficácia das vacinas COVID-19, até o momento, não há estudos publicados atuais sobre a relação entre a microbiota intestinal-pulmonar e a vacinação para essa infecção. Apesar disso, pesquisadores em todo o mundo têm trabalhado continuamente na busca de vacinas contra COVID-19 e algumas delas foram recentemente aprovadas para uso humano ( 140 - 142 ).
Diante do conhecimento atual, a modulação da microbiota está sendo investigada como uma possível terapia adjuvante para COVID-19. D'Ettorre et al. ( 143 ) avaliaram o impacto dos probióticos na redução da progressão da doença, em 28 pacientes. Os sujeitos incluídos apresentavam febre, acometimento pulmonar e solicitaram oxigenoterapia não invasiva. Os pacientes receberam antibióticos, tocilizumabe e hidroxicloroquina, isoladamente ou em combinação, e a administração de probióticos multistrain (2.400 bilhões de bactérias / dia). A formulação probiótica continha Lactobacillus acidophilus , L. helveticus , L. paracasei , L. plantarum , L. brevis , Bifidobacterium lactis eStreptococcus thermophilus. Após 3 dias de suplementação, todos os pacientes do grupo probiótico apresentaram remissão da diarreia e resolução dos demais sintomas, quando comparados a 42 controles saudáveis. Após 7 dias, o grupo probiótico apresentou redução significativa no risco estimado de insuficiência respiratória, e nas internações em unidades de terapia intensiva e mortalidade, apontando para o importante papel do eixo intestino-pulmão no controle da SARS-CoV-2 infecção ( 143 ). Além disso, no momento em que escrevemos esta revisão, há aproximadamente 10 ensaios clínicos registrados no ClinicalTrials.gov e atualmente em andamento para avaliar o impacto do uso de probióticos e moduladores da microbiota intestinal no COVID-19. tabela 1resume os principais aspectos desses ensaios clínicos em pacientes COVID-19.

Tabela 1 Ensaios clínicos em andamento que testam a eficácia da modulação da microbiota intestinal em pacientes com COVID-19 (ClinicalTrials.gov).
Um estudo piloto aberto, prospectivo, tipo caso-controle, conduzido no Hospital Universitario del Vinalopó, Espanha, tem como objetivo avaliar o efeito da administração oral diária de uma mistura de probióticos, durante 30 dias, na melhora dos sintomas, dias de internação e eliminação do vírus em 40 pacientes COVID-19 (NCT04390477). Um ensaio clínico randomizado simples-cego no Canadá, no Centre hospitalier de l'Université de Montréal, está planejando examinar o impacto da administração intranasal duas vezes ao dia com Lactococcus lactis W136, durante 14 dias, em 40 pacientes com COVID-19 não hospitalizados (NCT04458519). Além disso, um ensaio clínico multicêntrico, randomizado, quádruplo-cego, investigará o efeito preventivo da administração oral de Lactobacillus Coryniformis K8 (3 x 10 9UFC / dia) em 314 profissionais de saúde expostos ao COVID-19 durante 2 meses (NCT04366180).
No Hospital Geral Dr. Manuel Gea Gonzalez, México, um ensaio clínico randomizado controlado avaliará a segurança e eficácia da suplementação oral diária com algumas cepas de Lactobacillus plantarum e Pediococcus acidilactici CECT 7483, durante 30 dias, em 300 pacientes com COVID-19 leve . Os objetivos deste estudo são avaliar o risco de progressão para doença moderada / grave ou óbito, além de investigar a frequência e gravidade dos sintomas gastrointestinais, o envolvimento pulmonar, a carga viral e a modulação da microbiota fecal em correlação com as melhorias clínicas (NCT04517422). Na Áustria, na Medical University of Graz, um estudo randomizado quádruplo-cego e controlado por placebo investigará os efeitos do tratamento oral simbiótico comBifidobacterium bifidum , B. lactis , Enterococcus faecium , Lactobacillus acidophilus , L. paracasei , L. plantarum , L. rhamnosus , L. salivarius , inulina e frutooligossacarídeo (FOS) em 108 voluntários durante 30 dias. Os pesquisadores levantaram a hipótese de que a terapia simbiótica poderia diminuir a duração da diarreia, melhorar a consistência das fezes, inflamação intestinal, disbiose e os sintomas gastrointestinais de COVID-19 (NCT04420676). Nos Estados Unidos, um estudo randomizado, triplo-cego e controlado por placebo está sendo conduzido na Duke University para avaliar o efeito da ingestão oral diária de cápsulas contendo Lactobacillus rhamnosus GG na microbiota intestinal em 1.000 contatos domiciliares expostos de pacientes com COVID-19, durante 28 dias (NCT04399252).
O papel dos probióticos e outras estratégias com o objetivo de modular a microbiota intestinal em COVID-19 precisam de mais investigações, especialmente ensaios clínicos randomizados, duplo-cegos e controlados, incluindo coortes maiores em diferentes idades e cursos da doença ( 64 ). Nem todos os probióticos devem ser usados para conter infecções respiratórias, e as variações dos estudos incluem diferenças em cepas específicas, duração da suplementação, formas de administração, doses e tempos de acompanhamento ( 64 , 102 ). Finalmente, uma vez que COVID-19 é essencialmente uma doença inflamatória, a formação da microbiota intestinal do pulmão pelo uso de probióticos ( Figura 1) poderia representar uma importante ferramenta auxiliar no controle da inflamação excessiva que geralmente culmina no pior prognóstico da doença.
Conclusões
As superfícies mucosas, como nos pulmões e no intestino, desempenham um papel essencial na modulação das respostas imunológicas, combatendo microorganismos patogênicos e evitando inflamação excessiva ou danos aos tecidos. Este ajuste fino da imunidade local também depende do equilíbrio da microbiota local, enquanto uma quebra na tolerância da mucosa juntamente com uma condição disbiótica podem favorecer o estabelecimento e progressão de infecções, como a causada pelo vírus SARS-CoV-2 . Além disso, uma vez que em COVID-19 as mucosas respiratórias e gastrointestinais são afetadas, juntamente com alterações relevantes na microbiota local e inflamação, é plausível supor que as terapias adjuvantes baseadas na modulação do eixo intestino-pulmão e no restabelecimento da eubiose poderiam ser uma abordagem terapêutica importante para restringir as consequências prejudiciais da COVID-19. No entanto, mais estudos ainda são necessários para desvendar a eficácia dessas intervenções de base microbiana, especialmente em casos graves de COVID-19.
Contribuições do autor
GO desenhou o estudo e escreveu e revisou o manuscrito. CO, CP e LS escreveram o manuscrito. CC projetou, escreveu e revisou o estudo. Todos os autores contribuíram com o artigo e aprovaram a versão submetida.
Financiamento
Este trabalho foi apoiado pela CAPES (Código Financeiro 001), CNPq (310174 / 2016-3 e 309583 / 2019-5), e Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP 2017 / 08651.1 e 2021 / 00229-4) .
Conflito de interesses
Os autores declaram que a pesquisa foi realizada na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Agradecimentos
Agradecemos o apoio institucional da UNESP, USP e dos órgãos financeiros que contribuíram com o estudo.
Referências
1. Donaldson GP, Lee SM, Mazmanian SK. Biogeografia intestinal da microbiota bacteriana. Nat Rev Microbiol (2016) 14: 20–32. doi: 10.1038 / nrmicro3552
2. Li M, Wang B, Zhang M, Rantalainen M, Wang S, Zhou H, et al. Micróbios intestinais simbióticos modulam os fenótipos metabólicos humanos. Proc Natl Acad Sci (2008) 105: 2117–22. doi: 10.1073 / pnas.0712038105
3. Abrahamsson TR, Jakobsson HE, Andersson AF, Björkstén B, Engstrand L, Jenmalm MC. A diversidade da microbiota intestinal baixa na primeira infância precede a asma na idade escolar. Clin Exp Allergy (2014) 44: 842–50. doi: 10.1111 / cea.12253
4. Dickson RP, Singer BH, Newstead MW, Falkowski NR, Erb-Downward JR, Standiford TJ, et al. Enriquecimento do microbioma pulmonar com bactérias intestinais na sepse e na síndrome do desconforto respiratório agudo. Nat Microbiol (2016) 1: 16113. doi: 10.1038 / nmicrobiol.2016.113
5. He Y, Wen Q, Yao F, Xu D, Huang Y, eixo Wang J. Gut-pulmão: As contribuições microbianas e implicações clínicas. Crit Rev Microbiol (2017) 43: 81–95. doi: 10.1080 / 1040841X.2016.1176988
6. Trompette A, Gollwitzer ES, Yadava K., Sichelstiel AK, Sprenger N, Ngom-Bru C, et al. O metabolismo da fibra alimentar na microbiota intestinal influencia as doenças alérgicas das vias aéreas e a hematopoiese. Nat Med (2014) 20: 159–66. doi: 10.1038 / nm.3444
7. Wang H, Dai W, Feng X, Zhou Q, Wang H, Yang Y, et al. A composição da microbiota no trato respiratório superior de crianças saudáveis em Shenzhen, China, difere dos locais e idades respiratórias. BioMed Res Int (2018) 2018: 6515670. doi: 10.1155 / 2018/6515670
8. Groves HT, Cuthbertson L, James P, Moffatt MF, Cox MJ, Tregoning JS. Doença respiratória após infecção pulmonar viral altera a microbiota intestinal murina. Front Immunol (2018) 9: 182. doi: 10.3389 / fimmu.2018.00182
9. Wang J, Li F, Wei H, Lian ZX, Sun R, Tian Z. A infecção pelo vírus influenza respiratório induz lesão imunológica intestinal por meio de inflamação dependente de células Th17 mediada por microbiota. J Exp Med (2014) 211: 2397–410. doi: 10.1084 / jem.20140625
10. Budden KF, Gellatly SL, Wood DLA, Cooper MA, Morrison M., Hugenholtz P, et al. Ligações patogênicas emergentes entre a microbiota e o eixo intestino-pulmão. Nat Rev Microbiol (2017) 15: 55–63. doi: 10.1038 / nrmicro.2016.142
11. Levy M, Kolodziejczyk AA, Thaiss CA, Elinav E. Dysbiosis and the immun system. Nat Rev Immunol (2017) 17: 219–32. doi: 10.1038 / nri.2017.7
12. Al Nabhani Z, Eberl G. Impressão do sistema imunológico pela microbiota no início da vida. Mucosal Immunol (2020) 13: 183–9. doi: 10.1038 / s41385-020-0257-y
13. Dang AT, Marsland BJ. Micróbios, metabólitos e o eixo intestino-pulmão. Mucosal Immunol (2019) 12: 843–50. doi: 10.1038 / s41385-019-0160-6
14. Zhang D, Li S, Wang N, Tan HY, Zhang Z, Feng Y. A conversa cruzada entre a microbiota intestinal e os pulmões nas doenças pulmonares comuns. Front Microbiol (2020) 11: 301. doi: 10.3389 / fmicb.2020.00301
15. Trompette A, Gollwitzer ES, Pattaroni C, Lopez-Mejia IC, Riva E, Pernot J, et al. A fibra dietética confere proteção contra a gripe por meio da hematopoiese de monócitos de patrulhamento de Ly6c- e do metabolismo de células T CD8 +. Immunity (2018) 48: 992–1005. doi: 10.1016 / j.immuni.2018.04.022
16. Ichinohe T, Pang IK, Kumamoto Y, Peaper DR, Ho JH, Murray TS, et al. A microbiota regula a defesa imunológica contra a infecção pelo vírus influenza A do trato respiratório. Proc Natl Acad Sci USA (2011) 108: 5354–9. doi: 10.1073 / pnas.1019378108
17. Prompetchara E, Ketloy C, Palaga T. Respostas imunológicas em COVID-19 e vacinas potenciais: Lições aprendidas com a epidemia de SARS e MERS. Asian Pac J Allergy Immunol (2020) 38: 1-9. doi: 10.12932 / AP-200220-0772
18. Gensollen T, Iyer SS, Kasper DL, Blumberg RS. Como a colonização pela microbiota no início da vida molda o sistema imunológico. Science (2016) 352: 539–44. doi: 10.1126 / science.aad9378
19. Bain CC, Cerovic V. Interações da microbiota com o sistema imunológico da mucosa. Immunology (2020) 159: 1-3. doi: 10.1111 / imm.13159
20. Yang X, Yu Y, Xu J, Shu H, Xia J, Liu H, et al. Curso clínico e resultados de pacientes criticamente enfermos com pneumonia SARS-CoV-2 em Wuhan, China: um estudo observacional, retrospectivo e centralizado. Lancet Respir Med (2020) 8: 475–81. doi: 10.1016 / S2213-2600 (20) 30079-5
21. Agarwal A, Chen A, Ravindran N, To C, Thuluvath PJ. Manifestações gastrointestinais e hepáticas de COVID-19. J Clin Exp Hepatol (2020) 10: 263–5. doi: 10.1016 / j.jceh.2020.03.001
22. Lamers MM, Beumer J, van der Vaart J., Knoops K., Puschhof J, Breugem TI, et al. O SARS-CoV-2 infecta de forma produtiva os enterócitos intestinais humanos. Science (2020) 369: 50–4. doi: 10.1126 / science.abc1669
23. Leung WK, To KF, Chan PKS, Chan HLY, Wu AKL, Lee N, et al. Envolvimento entérico de infecção por coronavírus associada à síndrome respiratória aguda grave. Gastroenterology (2003) 125: 1011–7. doi: 10.1016 / s0016-5085 (03) 01215-0
24. Ettayebi K, Crawford SE, Murakami K., Broughman JR, Karandikar U, Tenge VR, et al. Replicação de norovírus humanos em enteróides humanos derivados de células-tronco. Science (2016) 353: 1387–93. doi: 10.1126 / science.aaf5211
25. Crawford SE, Ramani S, Tate JE, Parashar UD, Svensson L, Hagbom M, et al. Infecção por rotavírus. Nat Rev Dis Primer (2017) 3: 17083. doi: 10.1038 / nrdp.2017.83
26. Cheung KS, Hung IFN, Chan PPY, Lung KC, Tso E, Liu R, et al. Manifestações gastrointestinais de infecção por SARS-CoV-2 e carga de vírus em amostras fecais de uma coorte de Hong Kong: revisão sistemática e meta-análise. Gastroenterology (2020) 159: 81–95. doi: 10.1053 / j.gastro.2020.03.065
27. Lin L, Jiang X, Zhang Z, Huang S, Zhang Z, Fang Z, et al. Sintomas gastrointestinais de 95 casos com infecção por SARS-CoV-2. Gut (2020) 69: 997–1001. doi: 10.1136 / gutjnl-2020-321013
28. Jin X, Lian JS, Hu JH, Gao J, Zheng L, Zhang YM, et al. Características epidemiológicas, clínicas e virológicas de 74 casos de doença infectada por coronavírus 2019 (COVID-19) com sintomas gastrointestinais. Gut (2020) 69: 1002–9. doi: 10.1111 / irv.12758
29. Guan WJ, Ni ZY, Hu Y, Liang WH, Ou CQ, He JX, et al. Características Clínicas da Doença do Coronavírus 2019 na China. N Engl J Med (2020) 382: 1708–20. doi: 10.1056 / NEJMoa2002032
30. Pan L, Mu M, Yang P, Sun Y, Wang R, Yan J, et al. Características clínicas de pacientes com COVID-19 com sintomas digestivos em Hubei, China: um estudo descritivo, transversal e multicêntrico. Am J Gastroenterol (2020) 115: 766–73. doi: 10.14309 / ajg.0000000000000620
31. Xiao F, Tang M, Zheng X, Liu Y, Li X, Shan H. Evidence for Gastrointestinal Infection of SARS-CoV-2. Gastroenterology (2020) 158: 1831–3. doi: 10.1053 / j.gastro.2020.02.055
32. Xiao F, Sun J, Xu Y, Li F, Huang X, Li H, et al. SARS-CoV-2 infeccioso em fezes de paciente com COVID-19 grave. Emerg Infect Dis (2020) 26: 1920–2. doi: 10.3201 / eid2608.200681
33. Chen Y, Chen L, Deng Q, Zhang G, Wu K, Ni L, et al. A presença de RNA SARS-CoV-2 nas fezes de pacientes com COVID-19. J Med Virol (2020) 92: 833–40. doi: 10.1002 / jmv.25825
34. Ong J, Young BE, Ong S. COVID-19 em gastroenterologia: uma perspectiva clínica. Gut (2020) 69: 1144–5. doi: 10.1136 / gutjnl-2020-321051
35. Wu Y, Guo C, Tang L, Hong Z, Zhou J, Dong X, et al. Presença prolongada de RNA viral SARS-CoV-2 em amostras fecais. Lancet Gastroenterol Hepatol (2020) 5: 434-5. doi: 10.1016 / S2468-1253 (20) 30083-2
36. Xu Y, Li X, Zhu B, Liang H, Fang C, Gong Y, et al. Características da infecção pediátrica por SARS-CoV-2 e possíveis evidências de eliminação persistente do vírus fecal. Nat Med (2020) 26: 502-5. doi: 10.1038 / s41591-020-0817-4
37. Wang W, Xu Y, Gao R, Lu R, Han K, Wu G, et al. Detecção de SARS-CoV-2 em diferentes tipos de espécimes clínicos. JAMA (2020) 323: 1843–4. doi: 10.1001 / jama.2020.3786
38. Gaebler C, Wang Z, Lorenzi JCC, Muecksch F, Finkin S, Tokuyama M, et al. Evolution of Antibody Immunity to SARS-CoV-2 (2020). Disponível em: https://www.biorxiv.org/content/10.1101/2020.11.03.367391v2 (Acessado em 15 de janeiro de 2021).
39. Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. Surto de pneumonia associado a um novo coronavírus de provável origem em morcego. Nature (2020) 579: 270-3. doi: 10.1038 / s41586-020-2012-7
40. Wan Y, Shang J, Graham R, Baric RS, Li F. Receptor Recognition by the Novel Coronavirus de Wuhan: an Analysis Based on Decade-Long Structural Studies of SARS Coronavirus. J Virol (2020) 94: e00127–20. doi: 10.1128 / JVI.00127-20
41. Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, et al. A entrada na célula SARS-CoV-2 depende de ACE2 e TMPRSS2 e é bloqueada por um inibidor de protease clinicamente comprovado. Cell (2020) 181: 271–80. doi: 10.1016 / j.cell.2020.02.052
42. Hamming I, Timens W, Bulthuis MLC, Lely AT, Navis GJ, van Goor H. Distribuição no tecido da proteína ACE2, o receptor funcional para o coronavírus SARS. Um primeiro passo para entender a patogênese da SARS. J Pathol (2004) 203: 631–7. doi: 10.1002 / caminho.1570
43. Zhang H, Kang Z, Gong H, Xu D, Wang J, Li Z, et al. O sistema digestivo é uma rota potencial de COVID-19: uma análise do padrão de coexpressão de uma única célula de proteínas-chave no processo de entrada viral. Gut (2020) 69: 1010–8. doi: 10.1136 / gutjnl-2020-320953
44. Stanifer ML, Kee C, Cortese M, Zumaran CM, Triana S, Mukenhirn M, et al. Papel crítico do interferon tipo III no controle da infecção por SARS-CoV-2 em células epiteliais intestinais humanas. Cell Rep (2020) 32: 107863. doi: 10.1016 / j.celrep.2020.107863
45. Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, et al. Características epidemiológicas e clínicas de 99 casos de novos casos de pneumonia por coronavírus em Wuhan, China: um estudo descritivo. Lancet (2020) 395: 507–13. doi: 10.1016 / S0140-6736 (20) 30211-7
46. Schmulson M, Dávalos MF, Berumen J. Cuidado: Os sintomas gastrointestinais podem ser uma manifestação de COVID-19. Rev Gastroenterol Mex (2020) 85: 282–7. doi: 10.1016 / j.rgmx.2020.04.001
47. Konturek PC, Harsch IA, Neurath MF, Zopf Y. COVID-19 - mais do que doença respiratória: a perspectiva de um gastroenterologista. J Physiol Pharmacol (2020) 71 (2): 179–89. doi: 10.26402 / jpp.2020.2.02
48. Xiong XL, Wong KK-Y, Chi SQ, Zhou AF, Tang JQ, Zhou LS, et al. Estudo comparativo das características clínicas e tendência epidemiológica de 244 crianças infectadas com COVID-19 com ou sem sintomas gastrointestinais. Gut (2021) 70: 436–8. doi: 10.1136 / gutjnl-2020-321486
49. Liang W, Feng Z, Rao S, Xiao C, Xue X, Lin Z, et al. A diarreia pode estar subestimada: um elo que faltava no novo coronavírus de 2019. Gut (2020) 69: 1141–3. doi: 10.1136 / gutjnl-2020-320832
50. Xu H, Zhong L, Deng J, Peng J, Dan H, Zeng X, et al. Alta expressão do receptor ACE2 de 2019-nCoV nas células epiteliais da mucosa oral. Int J Oral Sci (2020) 12: 8. doi: 10.1038 / s41368-020-0074-x
51. Hashimoto T, Perlot T, Rehman A, Trichereau J, Ishiguro H, Paolino M, et al. ACE2 liga a desnutrição de aminoácidos à ecologia microbiana e à inflamação intestinal. Nature (2012) 487: 477–81. doi: 10.1038 / nature11228
52. Dethlefsen L, Relman DA. Recuperação incompleta e respostas individualizadas da microbiota intestinal distal humana à perturbação antibiótica repetida. Proc Natl Acad Sci USA (2011) 108 (Suplemento 1): 4554–61. doi: 10.1073 / pnas.1000087107
53. Willing BP, Russell SL, Finlay BB. Mudando o equilíbrio: efeitos dos antibióticos no mutualismo hospedeiro-microbiota. Nat Rev Microbiol (2011) 9: 233–43. doi: 10.1038 / nrmicro2536
54. Dhar D, Mohanty A. Gut microbiota e Covid-19- possível link e implicações. Virus Res (2020) 285: 198018. doi: 10.1016 / j.virusres.2020.198018
55. Abt MC, Osborne LC, Monticelli LA, Doering TA, Alenghat T, Sonnenberg GF, et al. Bactérias comensais calibram o limite de ativação da imunidade antiviral inata. Immunity (2012) 37: 158–70. doi: 10.1016 / j.immuni.2012.04.011
56. Ganal SC, Sanos SL, Kallfass C., Oberle K., Johner C., Kirschning C., et al. O priming de células natural killer por fagócitos mononucleares não mucosos requer sinais instrutivos da microbiota comensal. Immunity (2012) 37: 171–86. doi: 10.1016 / j.immuni.2012.05.020
57. Brown EM, Kenny DJ, Xavier RJ. Regulação da microbiota intestinal de células T durante a inflamação e autoimunidade. Annu Rev Immunol (2019) 37: 599–624. doi: 10.1038 / s41467-017-01803-x
58. Haak BW, Littmann ER, Chaubard JL, Pickard AJ, Fontana E, Adhi F, et al. Impacto da colonização intestinal com microbiota produtora de butirato na infecção viral respiratória após alo-HCT. Blood (2018) 131: 2978–86. doi: 10.1182 / blood-2018-01-828996
59. Yildiz S, Mazel-Sanchez B, Kandasamy M, Manicassamy B, Schmolke M. Influenza A infecção pelo vírus impacta a dinâmica da microbiota sistêmica e causa disbiose entérica quantitativa. Microbiome (2018) 6: 9. doi: 10.1186 / s40168-017-0386-z
60. Bradley KC, Finsterbusch K., Schnepf D, Crotta S, Llorian M, Davidson S, et al. Os sinais de interferon tônico impulsionados pela microbiota nas células do estroma pulmonar protegem da infecção pelo vírus da gripe. Cell Rep (2019) 28: 245–256.e4. doi: 10.1016 / j.celrep.2019.05.105
61. Neyt K, Lambrecht BN. O papel dos subconjuntos de células dendríticas do pulmão na imunidade aos vírus respiratórios. Immunol Rev (2013) 255: 57–67. doi: 10.1111 / imr.12100
62. Stefan KL, Kim MV, Iwasaki A, Kasper DL. Modulação da microbiota comensal da resistência natural à infecção por vírus. Cell (2020) 183: 1312–24.e10. doi: 10.1016 / j.cell.2020.10.047
63. Shokri S, Mahmoudvand S, Taherkhani R, Farshadpour F. Modulação da resposta imune pelo coronavírus da síndrome respiratória do Oriente Médio. J Cell Physiol (2019) 234: 2143 ± 51. doi: 10.1002 / jcp.27155
64. Villena J, Kitazawa H. A modulação da imunidade antiviral da mucosa por imunobióticos: eles poderiam oferecer algum benefício na pandemia de SARS-CoV-2? Front Physiol (2020) 11: 699. doi: 10.3389 / fphys.2020.00699
65. Kitazawa H, Villena J. Modulation of Respiratory TLR3-Anti-Viral Response by Probiotic Microorganisms: Lessons Learned from Lactobacillus rhamnosus CRL1505. Front Immunol (2014) 5: 201. doi: 10.3389 / fimmu.2014.00201
66. Grayson MH, Camarda LE, Hussain S-RA, Zemple SJ, Hayward M, Lam V, et al. A ruptura da microbiota intestinal reduz as células T regulatórias e aumenta a mortalidade por infecções virais respiratórias por meio do aumento da produção de IFNγ. Front Immunol (2018) 9: 1587. doi: 10.3389 / fimmu.2018.01587
67. Zelaya H, Alvarez S, Kitazawa H, Villena J. Respiratory Antiviral Immunity and Immunobiotics: Benéfico Effects on Inflammation-Coagulation Interaction during Influenza Virus Infection. Front Immunol (2016) 7: 633. doi: 10.3389 / fimmu.2016.00633
68. Clarke TB, Davis KM, Lysenko ES, Zhou AY, Yu Y, Weiser JN. O reconhecimento do peptidoglicano da microbiota pelo Nod1 aumenta a imunidade inata sistêmica. Nat Med (2010) 16: 228–31. doi: 10.1038 / nm.2087
69. Wang J, Zhao S, Liu M, Zhao Z, Xu Y, Wang P, et al. A expressão de ACE2 pelas células epiteliais do cólon está associada à infecção viral, imunidade e metabolismo energético (2020). Disponível em: https://www.medrxiv.org/content/10.1101/2020.02. 05.20020545v1 (acessado em 15 de janeiro de 2021).
70. Viana SD, Nunes S, Reis F. Desequilíbrio ACE2 como um jogador-chave para os maus resultados em pacientes COVID-19 com comorbidades relacionadas à idade - Papel da disbiose da microbiota intestinal. Aging Res Rev (2020) 62: 101123. doi: 10.1016 / j.arr.2020.101123
71. Xiong Y, Liu Y, Cao L, Wang D, Guo M, Jiang A, et al. Características transcriptômicas do fluido de lavagem broncoalveolar e células mononucleares do sangue periférico em pacientes com COVID-19. Emerg Microbes Infect (2020) 9: 761–70. doi: 10.1080 / 22221751.2020.1747363
72. Lucas C, Wong P, Klein J, Castro TBR, Silva J, Sundaram M, et al. As análises longitudinais revelam falha imunológica em COVID-19 grave. Nature (2020) 584: 463–9. doi: 10.1038 / s41586-020-2588-y
73. Mesoraca A, Margiotti K, Viola A, Cima A, Sparacino D, Giorlandino C. Avaliação de SARS-CoV-2 RNA viral em amostras fecais. Virol J (2020) 17:86. doi: 10.1186 / s12985-020-01359-1
74. Gupta S, Parker J, Smits S, Underwood J, Dolwani S. Verter viral persistente de SARS-CoV-2 nas fezes - uma revisão rápida. Colorretal Dis (2020) 22: 611–20. doi: 10.1111 / codi.15138
75. Gu S, Chen Y, Wu Z, Chen Y, Gao H, Lv L, et al. Alterações da microbiota intestinal em pacientes com influenza COVID-19 ou H1N1. Clin Infect Dis (2020) 4: ciaa709. doi: 10.1093 / cid / ciaa709
76. Villapol S. Gastrointestinal sintomas associados com COVID-19: impacto sobre o microbioma intestinal. Transl Res (2020) 226: 57–69. doi: 10.1016 / j.trsl.2020.08.004
77. Zuo T, Zhang F, Lui GCY, Yeoh YK, Li AYL, Zhan H, et al. Alterações na microbiota intestinal de pacientes com COVID-19 durante o período de hospitalização. Gastroenterology (2020) 159: 944–55.e8. doi: 10.1053 / j.gastro.2020.05.048
78. Zuo T, Zhan H, Zhang F, Liu Q, Tso EYK, Lui GCY, et al. Alterações no Microbioma Fúngico Fecal de Pacientes com COVID-19 Durante o Tempo de Internação até a Alta. Gastroenterology (2020) 159: 1302–10.e5. doi: 10.1053 / j.gastro.2020.06.048
79. Zuo T, Liu Q, Zhang F, Lui GC-Y, Tso EY, Yeoh YK, et al. Descrevendo a atividade viral fecal da SARS-CoV-2 em associação com a composição da microbiota intestinal em pacientes com COVID-19. Gut (2020) 70: 276–84. doi: 10.1136 / gutjnl-2020-322294
80. Budding A, Sieswerda E, Wintermans B, Bos M. An Age Dependent Pharyngeal Microbiota Signature Associated with SARS-CoV-2 Infection. Disponível em: https://papers.ssrn.com/sol3/papers.cfm?abstract_id=3582780 (Acessado em 15 de janeiro de 2021). Pré-impressões com a Lanceta.
81. Fan J, Li X, Gao Y, Zhou J, Wang S, Huang B, et al. A microbiota do tecido pulmonar é caracterizada por 20 pacientes falecidos com COVID-19. J Infect (2020) 81: e64–7. doi: 10.1016 / j.jinf.2020.06.047
82. Geva-Zatorsky N, Sefik E, Kua L, Pasman L, Tan TG, Ortiz-Lopez A, et al. Mining the Human Gut Microbiota for Immunomodulatory Organisms. Cell (2017) 168: 928–43.e11. doi: 10.1016 / j.cell.2017.01.022
83. Liu H, Zhu J, Hu Q, Rao X. Morganella morganii, um patógeno oportunista não negligente. Int J Infect Dis (2016) 50: 10–7. doi: 10.1016 / j.ijid.2016.07.006
84. De Maio F, Posteraro B, Ponziani FR, Cattani P, Gasbarrini A, Sanguinetti M. Nasopharyngeal Microbiota Profiling of SARS-CoV-2 Infected Patients. Biol Proced Online (2020) 22:18. doi: 10.1186 / s12575-020-00131-7
85. de Steenhuijsen Piters WAA, Heinonen S, Hasrat R, Bunsow E, Smith B, Suarez-Arrabal MC, et al. Microbiota nasofaríngea, transcriptoma do hospedeiro e gravidade da doença em crianças com infecção pelo vírus sincicial respiratório. Am J Respir Crit Care Med (2016) 194: 1104–15. doi: 10.1164 / rccm.201602-0220OC
86. Lee KH, Foxman B, Kuan G, López R, Shedden K, Ng S, et al. A microbiota respiratória: associações com sintomatologia da influenza e disseminação viral. Ann Epidemiol (2019) 37: 51–6.e6. doi: 10.1016 / j.annepidem.2019.07.013
87. Lee KH, Gordon A, Shedden K, Kuan G, Ng S, Balmaseda A, et al. O microbioma respiratório e a suscetibilidade à infecção pelo vírus influenza. PLoS One (2019) 14: e0207898. doi: 10.1371 / journal.pone.0207898
88. Edouard S, Million M, Bachar D, Dubourg G, Michelle C, Ninove L, et al. A microbiota nasofaríngea em pacientes com infecções virais do trato respiratório é enriquecida em patógenos bacterianos. Eur J Clin Microbiol Infect Dis (2018) 37: 1725–33. doi: 10.1007 / s10096-018-3305-8
89. Qin T, Geng T, Zhou H, Han Y, Ren H, Qiu Z, et al. Bactérias patobiônicas superdominantes na microbiota nasofaríngea como agentes causadores de infecção bacteriana secundária em pacientes com influenza. Emerg Microbes Infect (2020) 9: 605-15. doi: 10.1080 / 22221751.2020.1737578
90. Dickson RP, Erb-Downward JR, Freeman CM, McCloskey L, Beck JM, Huffnagle GB, et al. Variação Espacial no Microbioma Pulmonar Humano Saudável e o Modelo Ilha Adaptado de Biogeografia Pulmonar. Ann Am Thorac Soc (2015) 12: 821–30. doi: 10.1513 / AnnalsATS.201501-029OC
91. Morris A, Beck JM, Schloss PD, Campbell TB, Crothers K, Curtis JL, et al. Comparação do microbioma respiratório em não fumantes e fumantes saudáveis. Am J Respir Crit Care Med (2013) 187: 1067–75. doi: 10.1164 / rccm.201210-1913OC
92. Man WH, de Steenhuijsen Piters WAA, Bogaert D. A microbiota do trato respiratório: porteiro da saúde respiratória. Nat Rev Microbiol (2017) 15: 259–70. doi: 10.1038 / nrmicro.2017.14
93. Doi Y, Murray GL, Peleg AY. Acinetobacter baumannii: evolução das opções de tratamento de resistência antimicrobiana. Semin Respir Crit Care Med (2015) 36: 85–98. doi: 10.1055 / s-0034-1398388
94. Rajasingham R, Smith RM, Park BJ, Jarvis JN, Govender NP, Chiller TM, et al. Carga global da doença de meningite criptocócica associada ao HIV: uma análise atualizada. Lancet Infect Dis (2017) 17: 873–81. doi: 10.1016 / S1473-3099 (17) 30243-8
95. Gu J, Han B, Wang J. COVID-19: Gastrointestinal Manifestations and Potential Fecal-Oral Transmission. Gastroenterology (2020) 158: 1518–9. doi: 10.1053 / j.gastro.2020.02.054
96. Gao QY, Chen YX, Fang JY. Nova infecção por coronavírus e trato gastrointestinal. J Dig Dis (2019) 2020) 21: 125–6. doi: 10.1111 / 1751-2980.12851
97. He LH, Ren LF, Li JF, Wu YN, Li X, Zhang L. Flora intestinal como uma estratégia potencial para combater a infecção por SARS-CoV-2. Front Microbiol (2020) 11: 1388. doi: 10.3389 / fmicb.2020.01388
98. Luoto R, Ruuskanen O, Waris M, Kalliomäki M, Salminen S, Isolauri E. Suplementação de prebióticos e probióticos previne infecções por rinovírus em bebês prematuros: um ensaio randomizado controlado por placebo. J Allergy Clin Immunol (2014) 133: 405–13. doi: 10.1016 / j.jaci.2013.08.020
99. Waki N, Matsumoto M, Fukui Y, Suganuma H. Effects of probiótico Lactobacillus brevis KB290 na incidência de infecção por influenza entre escolares: um estudo piloto de rótulo aberto. Lett Appl Microbiol (2014) 59: 565–71. doi: 10.1111 / lam.12340
100. Namba K, Hatano M, Yaeshima T, Takase M, Suzuki K. Effects of Bifidobacterium longum BB536 administração na infecção por influenza, título de anticorpo de vacina de influenza e imunidade mediada por células em idosos. Biosci Biotechnol Biochem (2010) 74: 939–45. doi: 10.1271 / bbb.90749
101. Turner RB, Woodfolk JA, Borish L, Steinke JW, Patrie JT, Muehling LM, et al. Efeito do probiótico na resposta inflamatória inata e na eliminação viral na infecção experimental por rinovírus - um ensaio clínico randomizado. Benef Microbes (2017) 8: 207–15. doi: 10.3920 / BM2016.0160
102. Baud D, Dimopoulou Agri V, Gibson GR, Reid G, Giannoni E. Using Probiotics to Flatten the Curve of Coronavirus Disease COVID-2019 Pandemic. Front Public Health (2020) 8: 186. doi: 10.3389 / fpubh.2020.00186
103. Kumar RVJ, Seo BJ, Mun MR, Kim CJ, Lee I, Kim H, et al. Probiótico putativo Lactobacillus spp. do trato gastrointestinal suíno inibir coronavírus de gastroenterite transmissível e patógenos bacterianos entéricos. Trop Anim Health Prod (2010) 42: 1855–60. doi: 10.1007 / s11250-010-9648-5
104. Liu YS, Liu Q, Jiang YL, Yang WT, Huang HB, Shi CW, et al. O IFN-λ3 Porcino Exibido na Superfície em Lactobacillus plantarum Inibe a Infecção por Coronavírus Entérico Porcino de Células Epiteliais Intestinais Porcinas. J Microbiol Biotechnol (2020) 30: 515–25. doi: 10.4014 / jmb.1909.09041
105. Chai W., Burwinkel M., Wang Z, Palissa C., Esch B., Twardziok S., et al. Efeitos antivirais de uma cepa probiótica de Enterococcus faecium contra coronavírus de gastroenterite transmissível. Arch Virol (2013) 158: 799–807. doi: 10.1007 / s00705-012-1543-0
106. Wang K, Ran L, Yan T, Niu Z, Kan Z, Zhang Y, et al. Efeito da infecção por cepa anti-TGEV Miller do sobrenadante de Lactobacillus plantarum com base na via de sinalização JAK-STAT1. Front Microbiol (2019) 10: 2540. doi: 10.3389 / fmicb.2019.02540
107. Hill C, Guarner F, Reid G., Gibson GR, Merenstein DJ, Pot B, et al. Documento de consenso de especialistas. Declaração de consenso da Associação Científica Internacional de Probióticos e Prebióticos sobre o escopo e o uso apropriado do termo probiótico. Nat Rev Gastroenterol Hepatol (2014) 11: 506–14. doi: 10.1038 / nrgastro.2017.75
108. Gibson GR, Hutkins R, Sanders ME, Prescott SL, Reimer RA, Salminen SJ, et al. Documento de consenso de especialistas: Declaração de consenso da Associação Científica Internacional para Probióticos e Prebióticos (ISAPP) sobre a definição e o escopo dos prebióticos. Nat Rev Gastroenterol Hepatol (2017) 14: 491–502. doi: 10.1038 / nrgastro.2017.75
109. Swanson KS, Gibson GR, Hutkins R, Reimer RA, Reid G, Verbeke K, et al. Declaração de consenso da Associação Científica Internacional para Probióticos e Prebióticos (ISAPP) sobre a definição e o escopo dos simbióticos. Nat Rev Gastroenterol Hepatol (2020) 17 (11): 687–701. doi: 10.1038 / s41575-020-0344-2
110. Żółkiewicz J, Marzec A, Ruszczyński M, Feleszko W. Postbiotics-A Step Beyond Pre- and Probiotics. Nutrients (2020) 12: 2189. doi: 10.3390 / nu12082189
111. Wegh CAM, Geerlings SY, Knol J, Roeselers G, Belzer C. Postbiotics and their Potential Applications in Early Life Nutrition and Beyond. Int J Mol Sci (2019) 20: 4673. doi: 10.3390 / ijms20194673
112. Azad MAK, Sarker M, Wan D. Immunomodulatory Effects of Probiotics on Cytokine Profiles. BioMed Res Int (2018) 2018: 8063647. doi: 10.1155 / 2018/8063647
113. Zhang CX, Wang HY, Chen TX. Interações entre a Microflora Intestinal / Probióticos e o Sistema Imunológico. BioMed Res Int (2019) 2019: 6764919. doi: 10.1155 / 2019/6764919
114. Chiba Y, Shida K, Nagata S, Wada M, Bian L, Wang C, et al. Respostas de citocinas pró-inflamatórias bem controladas de células de patch de Peyer ao probiótico Lactobacillus casei. Immunology (2010) 130: 352–62. doi: 10.1111 / j.1365-2567.2009.03204.x
115. Christensen HR, Frøkiaer H, Pestka JJ. Os lactobacilos modulam diferencialmente a expressão de citocinas e marcadores de superfície de maturação em células dendríticas murinas. J Immunol (2002) 168: 171 ± 8. doi: 10.4049 / jimmunol.168.1.171
116. Lee A, Lee YJ, Yoo HJ, Kim M, Chang Y, Lee DS, et al. Consumo de Iogurte Lácteo Contendo Lactobacillus paracasei ssp. paracasei, Bifidobacterium animalis ssp. lactis e Lactobacillus plantarum tratado por calor melhora a função imunológica, incluindo a atividade natural das células assassinas. Nutrients (2017) 9: 558. doi: 10.3390 / nu9060558
117. Jeong M, Kim JH, Yang H, Kang SD, Song S, Lee D, et al. Lactobacillus plantarum KCTC 13314BP morto pelo calor aumenta a atividade fagocítica e os efeitos imunomoduladores por meio da ativação das vias MAPK e STAT3. J Microbiol Biotechnol (2019) 29: 1248–54. doi: 10.4014 / jmb.1905.05066
118. Arpaia N, Campbell C, Fan X, Dikiy S, van der Veeken J, deRoos P, et al. Os metabólitos produzidos por bactérias comensais promovem a geração de células T regulatórias periféricas. Nature (2013) 504: 451–5. doi: 10.1038 / nature12726
119. Kanmani P, Kim H. capacidades funcionais de cepas probióticas na atenuação da resposta inflamatória das células epiteliais intestinais induzida por estímulos TLR4. BioFactors (2019) 45: 223–35. doi: 10.1002 / biof.1475
120. Zheng B, van Bergenhenegouwen J, Overbeek S, van de Kant HJG, Garssen J, Folkerts G, et al. Bifidobacterium breve atenua a colite induzida por sulfato de sódio dextrano murino e aumenta as respostas de células T regulatórias. PLoS One (2014) 9: e95441. doi: 10.1371 / journal.pone.0095441
121. Sundararaman A, Ray M, Ravindra PV, Halami PM. Papel dos probióticos no combate às infecções virais com ênfase no COVID-19. Appl Microbiol Biotechnol (2020) 104: 8089–104. doi: 10.1007 / s00253-020-10832-4
122. Salva S, Villena J, Alvarez S. Atividade imunomodulatória de cepas de Lactobacillus rhamnosus isoladas de leite de cabra: impacto em infecções intestinais e respiratórias. Int J Food Microbiol (2010) 141: 82–9. doi: 10.1016 / j.ijfoodmicro.2010.03.013
123. Hori T, Kiyoshima J, Shida K, Yasui H. Efeito da administração intranasal de Lactobacillus casei Shirota na infecção pelo vírus da gripe do trato respiratório superior em ratos. Clin Diagn Lab Immunol (2001) 8: 593 ± 7. doi: 10.1128 / CDLI.8.3.593-597.2001
124. Chong HX, Yusoff NAA, Hor YY, Lew LC, Jaafar MH, Choi SB, et al. O Lactobacillus plantarum DR7 melhorou as infecções do trato respiratório superior por meio do aumento dos parâmetros imunológicos e inflamatórios: um estudo randomizado, duplo-cego e controlado por placebo. J Dairy Sci (2019) 102: 4783–97. doi: 10.3168 / jds.2018-16103
125. Hao Q, Dong BR, Wu T. Probióticos para prevenir infecções agudas do trato respiratório superior. Cochrane Database Syst Rev (2015) 2: CD006895. doi: 10.1002 / 14651858.CD006895.pub3
126. Bo L, Li J, Tao T, Bai Y, Ye X, Hotchkiss RS, et al. Probióticos para prevenir a pneumonia associada ao ventilador. Cochrane Database Syst Rev (2014) 10: CD009066. doi: 10.1002 / 14651858.CD009066.pub2
127. Su M, Jia Y, Li Y, Zhou D, Jia J. Probióticos para a Prevenção da Pneumonia Associada ao Ventilador: Uma Meta-Análise de Ensaios Controlados Randomizados. Respir Care (2020) 65: 673–85. doi: 10.4187 / respcare.07097
128. Pera A, Campos C, López N, Hassouneh F, Alonso C, Tarazona R, et al. Imunosenescência: Implicações para a resposta à infecção e vacinação em pessoas idosas. Maturitas (2015) 82 (1): 50–5. doi: 10.1016 / j.maturitas.2015.05.004
129. Amsterdam D, Ostrov BE. The Impact of the Microbiome on Immunosenescence. Immunol Invest (2018) 47 (8): 801–11. doi: 10.1080 / 08820139.2018.1537570
130. Guillemard E, Tondu F, Lacoin F, Schrezenmeir J. O consumo de um produto lácteo fermentado contendo o probiótico Lactobacillus casei DN-114001 reduz a duração das infecções respiratórias em idosos em um ensaio clínico randomizado. Br J Nutr (2010) 103 (1): 58–68. doi: 10.1017 / S0007114509991395
131. Szajewska H, Canani RB, Guarino A, Hojsak I, Indrio F, Kolacek S, et al. Probióticos para a prevenção da diarreia associada a antibióticos em crianças. J Pediatr Gastroenterol Nutr (2016) 62 (3): 495–506. doi: 10.1097 / MPG.0000000000001081
132. Lai HH, Chiu CH, Kong MS, Chang CJ, Chen CC. Probiótico Lactobacillus casei: eficaz para controlar a diarreia infantil por meio da alteração da microbiota intestinal e da atenuação dos marcadores inflamatórios fecais. Nutrients (2019) 11 (5): 1150. doi: 10.3390 / nu11051150
133. Szajewska H, Kołodziej M, Gieruszczak-Białek D, Skórka A, Ruszczyński M, Shamir R. Revisão sistemática com meta-análise: Lactobacillus rhamnosus GG para o tratamento de gastroenterite aguda em crianças - uma atualização de 2019. Aliment Pharmacol Ther (2019) 49 (11): 1376–1384. doi: 10.1111 / apt.15267
134. Guo Q, Goldenberg JZ, Humphrey C, El Dib R, Johnston BC. Probióticos para a prevenção da diarreia pediátrica associada a antibióticos. Cochrane Database Syst Rev (2019) 4: CD004827. doi: 10.1002 / 14651858.CD004827.pub5
135. Hanada S, Pirzadeh M, Carver KY, Deng JC. Alterações do Microbioma Induzidas por Infecção Viral Respiratória e Pneumonia Bacteriana Secundária. Front Immunol (2018) 9: 2640. doi: 10.3389 / fimmu.2018.02640
136. Levy M, Thaiss CA, Elinav E. Metabolites: mensageiros entre a microbiota e o sistema imunológico. Genes Dev (2016) 30 (14): 1589–97. doi: 10.1101 / gad.284091.116
137. Descamps HC, Herrmann B, Wiredu D, Thaiss CA. O caminho para o uso de metabólitos microbianos como terapias. EBioMedicine (2019) 44: 747–54. doi: 10.1016 / j.ebiom.2019.05.063
138. Lei H, Xu Y, Chen J, Wei X, Lam DM-K. Imunoproteção contra o vírus influenza H5N1 por administração oral de minicápsulas de Lactococcus lactis recombinante com revestimento entérico. Virology (2010) 407 (2): 319–24. doi: 10.1016 / j.virol.2010.08.007
139. Samuelson DR, Welsh DA, Shellito JE. Regulação da imunidade pulmonar e defesa do hospedeiro pela microbiota intestinal. Front Microbiol (2015) 6: 1085. doi: 10.3389 / fmicb.2015.01085
140. Ramasamy MN, Minassian AM, Ewer KJ, Flaxman AL, Folegatti PM, Owens DR, et al. Segurança e imunogenicidade da vacina ChAdOx1 nCoV-19 administrada em um regime de reforço inicial em adultos jovens e idosos (COV002): um ensaio clínico cego, randomizado, controlado, de fase 2/3. Lancet (2021) 396: 1979–93. doi: 10.1016 / S0140-6736 (20) 32466-1
141. Palacios R, Patiño EG, de Oliveira Piorelli R, Conde MTRP, Batista AP, Zeng G, et al. Ensaio clínico de fase III duplo-cego, randomizado e controlado por placebo para avaliar a eficácia e a segurança do tratamento de profissionais de saúde com a vacina adsorvida COVID-19 (inativada) fabricada pela Sinovac - PROFISCOV: um resumo estruturado de um protocolo de estudo para um controle randomizado tentativas. Trials (2020) 21: 853. doi: 10.1186 / s13063-020-04775-4
142. Polack FP, Thomas SJ, Kitchin N, Absalon J, Gurtman A, Lockhart S, et al. Segurança e eficácia da vacina Covid-19 de mRNA de BNT162b2. N Engl J Med (2020) 383: 2603-15. doi: 10.1056 / NEJMoa2034577
143. d'Ettorre G, Ceccarelli G, Marazzato M, Campagna G, Pinacchio C, Alessandri F, et al. Desafios no tratamento da infecção por SARS-CoV2: O papel da bacterioterapia oral como estratégia terapêutica complementar para evitar a progressão de COVID-19. Front Med (2020) 7: 389. doi: 10.3389 / fmed.2020.00389
Palavras-chave: COVID-19, inflamação, eixo intestino-pulmão, microbiota, probióticos
Citação: de Oliveira GLV, Oliveira CNS, Pinzan CF, de Salis LVV e Cardoso CRB (2021) Modulação da Microbiota do Eixo Intestino-Pulmão no COVID-19. Frente. Immunol. 12: 635471. doi: 10.3389 / fimmu.2021.635471
Recebido: 30 de novembro de 2020; Aceito: 18 de janeiro de 2021;
Publicado: 24 de fevereiro de 2021.
Revisados pela:
Yogesh Singh , Tübingen University Hospital, AlemanhaPam Kozlowski , Louisiana State University, Estados Unidos
Copyright © 2021 de Oliveira, Oliveira, Pinzan, de Salis e Cardoso. Este é um artigo de acesso aberto distribuído sob os termos da Creative Commons Attribution License (CC BY) . O uso, distribuição ou reprodução em outros fóruns é permitido, desde que o (s) autor (es) original (is) e o (s) proprietário (s) dos direitos autorais sejam creditados e que a publicação original nesta revista seja citada, de acordo com a prática acadêmica aceita. Não é permitida a utilização, distribuição ou reprodução em desacordo com estes termos.
* Correspondência: Cristina Ribeiro de Barros Cardoso, cristina@fcfrp.usp.br
Isenção de responsabilidade: Todas as reivindicações expressas neste artigo são exclusivamente dos autores e não representam necessariamente as de suas organizações afiliadas ou as do editor, dos editores e dos revisores. Qualquer produto que possa ser avaliado neste artigo ou alegação que possa ser feita por seu fabricante não é garantido ou endossado pelo editor.
Fonte:https://www.frontiersin.org/articles/10.3389/fimmu.2021.635471/full





Comentários
Postar um comentário